


БНБ
"ЭБНБ" (14252)
- Photogallery
- Естественные науки - Математика - Технология
- Авиация и машиностроение
- Высокие технологии
- Вычислительная техника
- Нанотехнология
- Роботехника
- Энергетика
- Электроника
Секвенирование по максаму-гилберту больших днк
Определение "Секвенирование по максаму-гилберту больших днк" в ЭБНБ
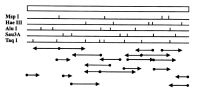 |
| Стратегия секвенирования |
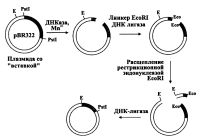 |
| Получение однонаправленных субклонов |
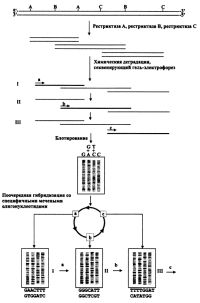 |
| Определение нуклеотидной последовательности |
Эта стратегия секвенирования ДНК методом химической деградации субклонированных фрагментов в векторах, содержащих полилинкерные последовательности, была довольно производительной в сравнении с бесполилинкерным вектором pBR322 и тем более по сравнению с применявшимся ранее подходом с элюцией рестриктазных фрагментов из геля. Поскольку метод химической деградации требовал довольно больших количеств ДНК, а элюция из полиакриламидного или агарозного гелей не очень эффективна, то разработка многочисленных векторов, несущих сайты для клонирования по многим рестрикционным эндонуклеазам, позволила, основываясь все на том же рестриктазном картировании, расщеплять вставку подходящими ферментами и получать необходимые субклоны в подобном векторе и затем их секвенировать в составе этого же вектора, получая меченные по одному концу фрагменты ДНК, как описано выше.
Другим преимуществом векторов, несущих полилинкерный участок, была довольно легкая возможность проводить дельта-рестрикционное субклонирование, также основанное на знании детальной рестриктазной карты. Сущность этого метода заключалась в использовании для укорочения вставки полилинкерных редкощепящих рестрикционных эндонуклеаз, сайты которых присутствовали и во вставке. Кроме этого, можно было применять с этой же целью ферменты, чьи сайты имелись во вставке, но отсутствовали в векторе. При этом сам вектор расщеплялся соответствующей полилинкерной рестрикционной эндонуклеазой, желательно образующей у линеаризуемого вектора со вставкой концы, совпадающие с таковыми при расщеплении вставки другим ферментом, что требовалось для облегчения самого этапа лигирования или упрощения подготовки к нему.
Усовершенствование плазмидных векторов для секвенирования ДНК методом химической деградации позволило создать еще более удобные специальные полилинкеры, содержащие в своей последовательности два участка расщепления рестрикционной эндонуклеазы 77A111I, имеющей сайт узнавания GACN^NNGTC [Eckert, 1987; Volckaert, 1987]. Удобство этих векторов заключалось в возможности получения с помощью данного фермента фрагментов ДНК, которые потом легко пометить только по одному концу, что как раз требуется для метода секвенирования ДНК по Максаму-Гилберту. Сайты полилинкерных рестрикционных эндонуклеаз, расположенные между фланкирующими сайтами рестрикционной эндонуклеазы ГАШИ, позволяли, если требовалось, проводить дельта-рестрикционное субклонирование.
Все рассмотренные выше стратегии секвенирования протяженных фрагментов ДНК методом Максама-Гилберта нельзя отнести к стратегиям случайного подхода, так как они основывались, как правило, на знании рестриктазной карты клонированного фрагмента ДНК и были рассчитаны на получение конкретных субфрагментов ДНК или субклонов. В то же время возможность случайного раздробления крупного фрагмента ДНК физическими методами и получение на их основе субклонов для их последующего секвенирования методом химической деградации принципиально возможны, хотя и не производительны. При этом, однако, необходимо отметить, что совершенствование векторных систем и их использование для определения нуклеотидных последовательностей протяженных фрагментов ДНК химической деградацией по Максаму-Гилберту не смогло значительно повысить производительность этого метода, разве что, кроме, метода мультиплексного секвенирования.
Первоначально разработанный для секвенирования ДНК по Максаму-Гилберту метод мультиплексного секвенирования ДНК, основанный на одновременном использовании целого набора плазмидных векторов plex00-20, позволял надеяться на резкое увеличение производительности этого метода секвенирования ДНК [Church, Kieffer-Higgins, 1988]. Так, двадцать независимо клонированных фрагментов ДНК, полученных в результате ультразвуковой обработки исходного фрагмента ДНК, в двадцати векторах plex после этапов совместного расщепления соответствующими рестрикционными эндонуклеазами и совместного проведения реакций химической деградации разделяли в одном секвенирующем геле и переносили на нейлоновый фильтр. Лестницы полос из всех двадцати клонов выявляли поочередной гибридизацией с мечеными праймерами, гомологичными каждый раз только для одного такого вектора из набора. Однако, несмотря на один общий этап химической деградации, позволяющий экономить время, метод мультиплексного секвенирования достаточно сложен, поскольку для дальнейшего выявления нуклеотидных последовательностей всех клонированных фрагментов ДНК необходим трудоемкий этап блот-гибридизации, удаление связанной метки и следующий этап гибридизации с другим праймером и так последовательно со всеми праймерами, гомологичными соответствующим участкам разных плазмид из набора plex00=20. В то же время нельзя не упомянуть о том, что значительная часть нуклеотидной последовательности полного генома архебактерии Methanobacterium thermoautotrophicum, составляющего более 1750 тпн, определена именно с помощью мультиплексного секвенирования ДНК путем химической деградации [Smith et al., 1997]. Этим же подходом было секвенировано около трети всего генома Mycoplasma capricolum, составляющего более 750 тпн и характеризующегося высоким (75%) содержанием АТ-пар [Dolan et al., 1995].
Ранее схожий метод мультиплексной геномной "прогулки" был применен для определения нуклеотидной последовательности неклонированного гена пируваткиназы Е. coli [Ohara et al., 1989]. Суть такой "прогулки", весьма напоминающей праймерную, заключается в расщеплении тотальной ДНК несколькими подходящими рестрикционными эндонуклеазами, разделении полученных фрагментов в денатурирующем секвенирующем гель-электрофорезе, их переносе на нейлоновый фильтр и многочисленных гибридизациях с мечеными олигонуклеотидами (рис.).
Первым таким меченым зондом служит синтезированный олигонуклеотид, комплементарный участку секвенируемой ДНК с известной последовательностью. Следующий, очередной зонд подбирают на основе уже определенной в предыдущем цикле нуклеотидной последовательности. Причем синтез такого олигонуклеотида, комплементарного той или иной цепи, позволяет осуществлять как секвенирование нового участка, так и подтверждающее секвенирование в обратном направлении.
| "ЭБНБ" >> "С" >> "СЕ" >> "СЕК" |
Статья про "Секвенирование по максаму-гилберту больших днк" в Энциклопедии БНБ была прочитана 4179 раз
| Коптим скумбрию в коробке |
| Буддийская молитва в Камбодже |
TOP 15
- Вши и борьба с ними
- Пижма обыкновенная
- Каштан конский обыкновенный
- Роль элементов в жизни растений
- Пшеница
- Иван-чай узколистный
- Календула лекарственная
- Обеззараживание химическими средствами
- Клевер луговой
- Дезинфекция текущая
- Душица обыкновенная
- Бессмертник песчаный
- Как устроены растения
- Морфологические признаки почвы
- Хвощ полевой